En esta unidad número 8 como en muchas otras
unidades que hemos vistos asido muy interesante, ya que pudimos entender los
procesos de la regulación de expresión genética, como se lleva a cabo, donde
ocurre, que factores intervienen en esos procesos, en cuál de los organismos es
más sencilla y donde es más complicado o complejo.
miércoles, 30 de mayo de 2012
8.3 REGULACIÓN DE LA TRANSCRIPCIÓN EN ORGANISMOS EUCARIÓTICOS.
Los
tipos de señales que pueden alterar la transcripción de un gen puede ser:
Señales hormonales:
PROTEÍNAS
QUE MODULAN LA TRANSCRIPCIÓN
En eucariotas, pueden actuar
como reguladores tanto moléculas de RNA como proteínas. Entre las proteínas,
las hay que forman parte de la holoenzima polimerasa (los factores de
transcripción), otras intervienen en la remodelación de la cromatina y un
tercer grupo se une al DNA para regular la transcripción, que es el que nos
ocupa.
1.- ACTIVADORES TRANSCRIPCIONALES
Los activadores son la
proteínas que se van a unir a los elementos distales (SDE y potenciadores) para
activar la transcripción.
2.- COACTIVADORES
Y CORREPRESORES
La acción de un activador de transcripción (o
de un represor) puede ejercerse directamente sobre el complejo basal (bien
sobre la RNA-polimerasa, alguno de los TFII o los TAFII), o a través de una
molécula intermediaria que puede ser un coactivador o un correpresor.
Se denomina coactivador si ayuda a activar la transcripción. Un mismo
coactivador puede recibir señales de distintos activadores para transmitirlos
hacia el complejo del promotor basal.
3.- TRANSACTIVADORES
Son aquellos que
directamente ejercen su acción interaccionando con el complejo de iniciación
formado en el promotor basal, bien sobre la propia polimerasa o, más
normalmente, sobre una de las TAF o de los TFII, para activar o reprimir la
transcripción, puesto que no son actividades excluyentes.
Potenciadores
La fuente de regulación más
potente es al de los elementos distales: los potenciadores (enhancers o
intensificadores). Su función es la de amplificar la transcripción del promotor
incluso más de 1000 veces.
Silenciamiento
de genes
La unión inespecífica de proteínas
reguladoras es un problema importante en los organismos con genomas grandes.
Para combatirla, los eucariotas han hecho que los genes tengan en torno a 5
dianas para proteínas reguladoras diferentes. Esta estrategia es útil para los
activadores de la transcripción porque es una estrategia eficiente y ahorra
esfuerzo. Una estrategia similar no es factible con los inhibidores de la
transcripción, por lo que se da poca regulación por silenciamiento.Inactivación mediante una proteína reguladora
Se consigue uniendo una proteína reguladora a cualquiera de los distintos elementos que forman los promotores.
PTGS: silenciamiento génico postranscripcional
Consiste en la degradación específica de los mRNA complementarios de una de las hebras del dsRNA. Los mRNA degradados suelen ser transcritos aberrantes de diversos orígenes. También se denomina cosupresión o extinción (quelling).
Silenciamiento por
metilación
No todos los organismos tienen el DNA
metilado. En los mamíferos, el DNA metilado forma heterocromatina a la que no
pueden acceder los factores de transcripción. Por tanto, los genes metilados no
se pueden transcribir ni tan siquiera residualmente. Se trata de un mecanismo
muy eficiente de silenciamiento génico que, además, disminuye la cantidad de
DNA que los factores de transcripción y la RNA-polimerasa tienen que rastrear
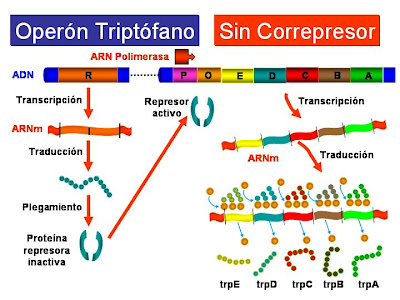
para buscar los promotores.8.4.2 OPERON DE TRIPTOFANO.
El
operón triptófano (operón trp) es un sistema de tipo reprensible, ya que el
aminoácido triptófano (Correpresor) impide la expresión de los genes necesarios
para su propia síntesis cuando hay niveles elevados de triptófano. Sin embargo,
en ausencia de triptófano o a niveles muy bajos se transcriben los genes del
operón trp. Los elementos del operón trp son en esencia semejantes a los del
operón lactosa:
·
Genes estructurales: existen cinco genes
estructurales en el siguiente orden trpE-trpD-trpC-trpB-trpA.
·
Elementos de control: promotor (P) y operador (O).
El promotor y el operador están al lado de los genes estructurales y en el
siguiente orden: P O trpE-trpD-trpC-trpB-trpA. Curiosamente, las enzimas
codificadas por estos cinco genes estructurales actúan en la ruta metabólica de
síntesis del triptófano en el mismo orden en el que se encuentran los genes en
el cromosoma.
·
Gen regulador (trpR): codifica para la proteína
reguladora. Este gen se encuentra en otra región del cromosoma bacteriano
aunque no muy lejos del operón.
·
Correpresor: triptófano.
OPERÓN
TRIPTÓFANO: EN AUSENCIA DE TRIPTÓFANO
En ausencia de triptófano, o cuando
hay muy poco, la proteína reguladora producto del gen trpR no es capaz de
unirse al operador de forma que la ARN-polimerasa puede unirse a la región promotora
y se transcriben los genes del operón triptófano.
OPERÓN
TRIPTÓFANO: EN PRESENCIA DE TRIPTÓFANO.
En presencia de
triptófano, el triptófano se une a la proteína reguladora o represora cambiando
su conformación, de manera que ahora si puede unirse a la región operadora y
como consecuencia la ARN-polimerasa no puede unirse a la región promotora y no
se transcriben los genes estructurales del operón trp.
Por tanto, la diferencia esencial
entre el operón lac (inducible) y el operón trp (represible), es que en este
último el represor del triptófano solamente es capaz de unirse al operador
cuando previamente está unido al trp.
Al estudiar más
profundamente el operón trp se encontró que además del mecanismo de regulación
represor-operador existía otro mecanismo de regulación que se denominó
regulación por atenuación.
BIBLIOGRAFÍA:
- www.itescam.edu.mx/principal/sylabus/fpdb/recursos/r46453.PDF
- antología de biología molecular.
8.2.1 OPERON DE LACTOSA (CONTROL POSITIVO).
Jacob, Monod y colaboradores analizaron el sistema de la lactosa en E.
coli, de manera que los resultados de sus estudios permitieron establecer el
modelo genético del Operón que permite comprender como tiene lugar la
regulación de la expresión génica en bacterias.
Un Operón es grupo de genes estructurales cuya
expresión está regulada por los mismos elementos de control (promotor y
operador) y genes reguladores.
LOS PRINCIPALES
ELEMENTOS QUE CONSTITUYEN UN OPERÓN SON LOS SIGUIENTES:
v Los genes
estructurales: llevan información para poli péptidos. Se
trata de los genes cuya expresión está regulada. Los operones bacterianos
suelen contener varios genes estructurales, son poligénicos o policistrónicos.
Hay algunos operones bacterianos que tienen un solo gene estructural. Los
operones eucarióticos suelen contener un sólo gen estructural siendo
monocistrónicos.
v El promotor
(P): se trata de un elemento de control que es
una región del ADN con una secuencia que es reconocida por la ARN polimerasa
para comenzar la transcripción. Se encuentra inmediatamente antes de los genes
estructurales. Abreviadamente se le designa por la letra P.
v El operador
(O): se trata de otro elemento de control que es
una región del ADN con una secuencia que es reconocida por la proteína
reguladora. El operador se sitúa entre la región promotora y los genes
estructurales. Abreviadamente se le designa por la letra O.
v El gen
regulador (i): secuencia de ADN que codifica para la
proteína reguladora que reconoce la secuencia de la región del operador. El gen
regulador está cerca de los genes estructurales del operón pero no está
inmediatamente al lado. Abreviadamente se le denomina gen i.
v Proteína
reguladora:
proteína codificada por el gen regulador. Está proteína se une a la región del
operador.
v Inductor:
sustrato o compuesto cuya presencia induce la expresión de los genes.
El Operón
lactosa, que abreviadamente se denomina Operón lac, es un sistema inducible que
está bajo control negativo, de manera que la proteína reguladora, producto del
gen regulador i, es un represor que impide la expresión de los genes
estructurales en ausencia del inductor. El inductor del sistema es la lactosa.
Como veremos más adelante, el operón lac también está bajo control positivo, ya
que existe otra proteína que estimula la
transcripción de los genes estructurales.
Los genes estructurales del operón lactosa son
los siguientes:
v El gen z+: codifica para
la βββ-galactosidasa que cataliza la
hidrolisis de la lactosa en glucosa más galactosa.
v El gen y+: codifica para
la galactósido permeasa que
transporta β-galactósidos al interior de
la célula bacteriana.
v El gen a+: codifica para
la tiogalactósido transacetilasa que cataliza la transferencia del grupo acetil
del acetil Coenzima A al 6-OH de un aceptor tiogalatósido. Este gen no está
relacionado con el metabolismo de la lactosa.
OPERÓN LACTOSA EN AUSENCIA DE LACTOSA
Sin embargo, en presencia del inductor (la lactosa), este se une a la
proteína reguladora que cambia su conformación y se suelta de la región
operadora dejando acceso libre a la ARN polimerasa para que se una a la región
promotora y se transcriban los genes estructurales. Por consiguiente, la
presencia del inductor hace que se expresen los genes estructurales del operón,
necesarios para metabolizar la lactosa.
OPERÓN LACTOSA EN PRESENCIA DE LACTOSA
También
es conveniente recordar que los tres genes
estructurales del operón lactosa se transcriben juntos en un mismo ARNm,
es decir que los ARN mensajeros de bacterias suelen ser policistrónicos, poligénicos o multigénicos.
Sin embargo, en eucariontes los mensajeros suelen ser monocistrónicos o mono génicos, es decir,
corresponden a la transcripción de un solo gen estructural.
OPERÓN LACTOSA: ARNM MULTIGÉNICO O POLICISTRÓNICO
BIBLIOGRAFIA:
- www.itescam.edu.mx/principal/sylabus/fpdb/recursos/r46453.PDF
- antología de biología molecular.
martes, 29 de mayo de 2012
8.2 REGULACIÓN DE LA TRANSCRIPCIÓN EN ORGANISMOS PROCARIÓTICOS.
La regulación de la síntesis de proteínas ocurre a nivel de la transcripción
y es una consecuencia de la interacción entre el ambiente químico de la
bacteria y proteínas codificadas por genes reguladores. Estas proteínas pueden
funcionar como controles negativos, reprimiendo la transcripción del mRNA, o
como controles positivos, intensificándola.
LA REGULACION DE LA TRANSCRIPCION EN ORGANISMOS
PROCARIOTICOS ESTA REGULADA POR LOS SIGUIENTES MECANISMOS:
CAMBIOS EN LA ESTRUCTURA DEL DNA
METILACIÓN:
La metilación
provoca un cambio de estructura en el apareamiento entre las bases nitrogenadas
que puede alterar su reconocimiento por algunas proteínas. El más conocido es
el de la metilasa dam que reconoce la secuencia GATC y metila la A.
La mayor parte de los genes cuya expresión se ve reprimida por la
metilación son genes cuya expresión sólo se necesita durante la replicación
(único momento en el que una cadena del DNA está transitoriamente
hemimetilado), permaneciendo reprimidos el resto del ciclo celular.
SUPERENROLLAMIENTO:
Para mantener una situación homeostática en la célula en relación al número
de superenrollamientos es necesario mantener con una regulación contraria los
genes topA (codifica la topoisomerasa I) y gyrA y gyrB (determinan las dos
subunidades de la DNA-topoisomerasa II). No se conot;; mso-fareast-language: ES;">Lo provocan aquellos cambios que, sin alterar ni la estructura del DNA ni
la de la RNA-polimerasa, sí que afectan la interacción entre ambas. Es
necesaria la comparecencia de una tercera molécula, habitualmente una proteína
(aunque a veces puede ser RNA). Tres mecanismos pueden alterar esta
interacción:
• Modificación de la
interacción entre RNA polimerasa y el promotor debido a la existencia de una
proteína reguladora y un efector.
• Modificación de la
interacción entre RNA polimerasa y el promotor debido a la existencia de una
proteína reguladora y un efector.
• Secuencias reguladoras a
distancia.
• Modificación de la terminación: anti
terminación.
BIBLIOGRAFIA:
Antología biología molecular
8.1 NIVELES DE REGULACIÓN DE LA EXPRESIÓN GENÉTICA.
A nivel de la regulación en
la expresión de los genes, La estrategia procariota pretende alcanzar
las máximas tasas de proliferación cuando el entorno se lo permita. En cambio,
la estrategia eucariota ha de ser distinta puesto que en los organismos
pluricelulares donde el medio intercelular es relativamente constante, el
control génico está al servicio de la especialización celular.
La genómica parece indicar que:
1.- el número de
genes no varía mucho entre las especies: los vertebrados tienen como mucho el
doble de genes que los invertebrados;
2.- el número de
genes no sirve para explicar la diversidad evoultiva por mutación o duplicación
génica;
3.- la
variabilidad de los genes se debe a la duplicación de genes en vez de la
creación de genes nuevos.
4.- la
complejidad evolutiva se correlaciona con el aumento de genes reguladores: en
las levaduras hay un gen regulador por cada 20 funcionales, pero en humanos hay
más de 3 000 reguladores para unos 30 000 genes.
NIVELES DE LA EXPRESIÓN GÉNICA EN EUCARIONTES
Las diferentes posibilidades de regulación de la expresión
génica en organismos eucariotas son:
I. Nivel de cromatina
II. Nivel transcripcional
III. Nivel postranscripcional
IV. Nivel traduccional
V. Nivel postraduccional
La regulación génica es un proceso muy complejo que puede llevarse a cabo a
varios niveles:
1.
Sobre la
cromatina: zonas transcripcionalmente activas vs. Zonas silentes
2.
Sobre la
transcripción: iniciación, sitios alternativos de iniciación, splicing.
Por eso, la complejidad de los organismos emerge de una regulación de la
expresión génica cada vez más elaborada y no de cambios o mutaciones en los
genes estructurales o enzimáticos: la secuencia de las proteínas se conserva
mucho a través de distintas especies, sin cambios importantes mientras que los
cambios en el orden de los elementos del promotor o de sus elementos
reguladores provocan alteraciones drásticas.
INTRODUCCIÓN, OBJETIVOS Y METODOLOGÍA.
INTRODUCCIÓN:
El mecanismo básico de como las bacterias
controlan su transcripción fue formulado por Jacob y Monod en 1961. Ellos
propusieron que la expresión de los genes está regulada por las interacciones
especificas que ocurren entre secuencias cis localizadas en el DNA con los
productos de secuencias codificadas en regiones trans. Los elementos en trans
son usualmente proteínas llamadas factores transcripcionales o proteínas
reguladoras, las cuales se sintetizan en respuesta a estímulos ambientales como
temperatura, osmolaridad, pH, o condiciones nocivas que evocan respuestas
generales de protección, como agentes que causan daño al DNA, así mismo, la presencia
de minerales, fuentes de energía, aceptores de electrones y metabolitos especıficos
constituyen una segunda clase de estímulos que modifican a la expresión genética.
La expresión genética es modulada en cada etapa
del inicio de la transcripción. De esta forma, los genes pueden encenderse o
apagarse de distintos modos. Por ejemplo, a través de modificaciones en la RNAP,
re arreglos en la estructura del DNA que conectan a los genes con promotores
particulares dada la acción de las proteínas reguladoras; pequeñas moléculas de
RNA que activan o inhiben la transcripción o finalmente modulando la elongación
y terminación de la transcripción en sitios especıficos. La mayoría de los estímulos
los detecta la célula a través de componentes solubles en el citoplasma ya sea
porque el estimulo logro penetrar en la célula, o bien porque el estimulo afecto
a algunos componentes celulares o del metabolismo que ahora pueden funcionar
directamente como pequeñas moléculas inductoras o co-represoras de las proteínas
reguladoras del inicio de la transcripción; logrando con ello, mediar una gran
parte de la respuesta celular.
BIBLIOGRAFIA:
smcg.ccg.unam.mx/...TranscripYRegulacion/GutierrezRM_Regulacion.
Integrar los Conocimientos anteriores con los mecanismos de regulación genética para entender a nivel molecular los procesos metabólicos.
METODOLOGIA:
La metodología que utilizare en esta unida número 8 como en las anteriores será publicando todos los trabajos, tareas y temas vistos en clase, en el tiempo que indique el profesor, apoyándome en la antología otorgada por el profesor de clase, además de páginas en internet, etc.
TEMARIO
|
|
TEMA
|
SUBTEMAS
|
|
VIII
|
REGULACION DE LA EXPRESION GENETICA.
|
8.1
NIVELES DE REGULACIÓN DE LA EXPRESIÓN GENÉTICA.
8.2
REGULACIÓN DE LA TRANSCRIPCIÓN EN ORGANISMOS PROCARIÓTICOS.
8.4.1
OPERON DE LACTOSA (CONTROL POSITIVO).
8.4.2
OPERON DE TRIPTOFANO (CONTROL NEGATIVO).
8.3
REGULACIÓN DE LA TRANSCRIPCIÓN EN ORGANISMOS EUCARIÓTICOS.
|
PORTADA
SEP SNEST DGEST
INSTITUTO TECNOLÓGICO DE CIUDAD ALTAMIRANO
PRESENTA:
ULISES SALMERON FARIAS
UNIDAD VIII
“REGULACION DE LA EXPRESION GENETICA”
No DE CONTROL: 09930082
CARRERA: LIC. EN BIOLOGIA
VI SEMESTRE
CIUDAD ALTAMIRANO GRO. MÉXICO A 29 DE MAYO DEL
2012
miércoles, 23 de mayo de 2012
7.4.1 MODIFICACIÓN DE PROTEÍNAS POSTRADUCCIÓN
La modificación postraduccional
de una proteina es un cambio químico ocurrido en
esta después de su sintesis proteica. Las modificaciones postraduccionales ocurren
mediante cambios químicos de los aminoacidos que constituyen las proteínas y
pueden ser de muchos tipos.
PLEGAMIENTO
Todavía no se sabe con certeza
cómo la información de una secuencia primaria de aminoácidos guía su estructura
tridimensional. El plegamiento comienza en cuanto se sintetizan más de 30 aa
—son los que el ribosoma protege—. Por ejemplo, la ß-galactosidasa comienza a
formar los tetrámeros antes de terminar cada monómero.
La
importancia del plegamiento se refleja en que algunas enfermedades tienen su
base molecular en proteínas mal plegadas:
- La enfermedad de Alzheimer se origina en la acumulación de la proteína
ß-amiloide mal plegada
- La enfermedad de las vacas locas, scrapie en ovejas o enfermedad de
Creutzfeld-Jacob se debe a la acumulación de la proteína priónica PrP
plegada incorrectamente que además cataliza la conversión de las PrP bien
plegada en una PrP mal plegada.
Pueden ocurrir
una vez finalizada la síntesis del péptido, una vez liberado del ribosoma o,
más frecuentemente, de forma simultánea a su síntesis. Son muchos los tipos de
modificaciones que se pueden encontrar.
AMINOÁCIDOS MODIFICABLES
Sin tener en
cuenta modificaciones del tipo entrecruzamiento, la unión de
fosfatidil-inositol, la formación de piroglutamato, la formación de diftamida,
la formación de al-lisina o la formación de dionas, los aminoácidos que no se
suelen modificar son Gly, Ala (aminoácidos pequeños), Leu, Ile, Val o Trp
(aminoácidos hidrófobos).
DESFORMILACIÓN
La desformilasa
procariótica elimina el formilo de la fMet en la primera posición de las
proteínas al poco de aparecer el extremo N fuera del ribosoma.
PUENTES DISULFURO
Se trata de la
formación de un enlace covalente entre dos Cys de la misma o distintas cadenas
polipeptídicas. Se consigue mediante una reacción redox catalizada por la proteína-disulfuro-isomerasa
en presencia de glutatión, que sufre el proceso inverso (ruptura de su doble
enlace). Si se revierte esta modificación, la proteína se desnaturaliza.
Suscribirse a:
Comentarios (Atom)